Cell Reports Methods:深圳湾实验室孙坤课题组揭示cfDNA片段化组学分析中的系统偏差
2024-06-14 测序中国 测序中国 发表于上海
该研究揭示了以参考基因组为基础的传统cfDNA分析流程中人种差异等因素所导致的系统偏差,并开发了一款无需参考基因组、快速、准确的cfDNA片段化组学分析算法Freefly。
近日,深圳湾实验室孙坤课题组在Cell Reports Methods期刊发表题为《Systematic biases in reference-based plasma cell-free DNA fragmentomic profiling》的文章。该研究揭示了以参考基因组为基础的传统cfDNA分析流程中人种差异等因素所导致的系统偏差,并开发了一款无需参考基因组、快速、准确的cfDNA片段化组学分析算法Freefly。

研究发现
01 cfDNA传统处理方法存在系统误差
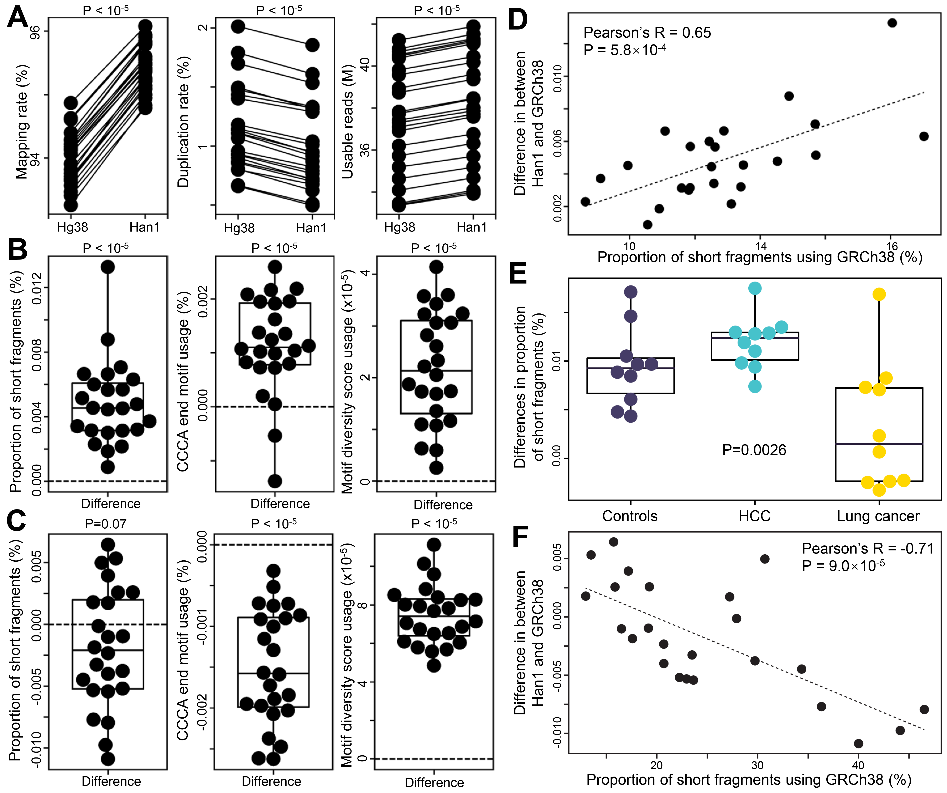
外周血循环游离DNA(circulating cell-free DNA,简称cfDNA)是一种细胞死亡后自然降解产生的DNA片段。cfDNA在癌症早期诊断、肿瘤起源追溯与分型、组织损伤监测等方向具有重要的意义。研究表明,肿瘤来源的cfDNA分子与背景cfDNA(主要来源于造血系统)存在显著的差异,例如片段大小、末端序列,是获得广泛验证的癌症检测标志物。在传统的分析流程中,为了获取cfDNA的片段大小和末端序列,需要将cfDNA测序数据比对到参考基因组上。该过程存在两个问题:一是参考基因组难以完美适配所有人种的基因序列,因此导致遗传背景依赖的偏差;二是所需要的时间较长,在一定程度上限制了cfDNA片段化组学在临床诊断中的实时应用。下图展示了在中国人群中,使用GRCh38(目前最常用的参考基因组)和Han1(来自中国南方人种)参考基因组得到的结果之间存在系统性偏差。
02 Freefly算法
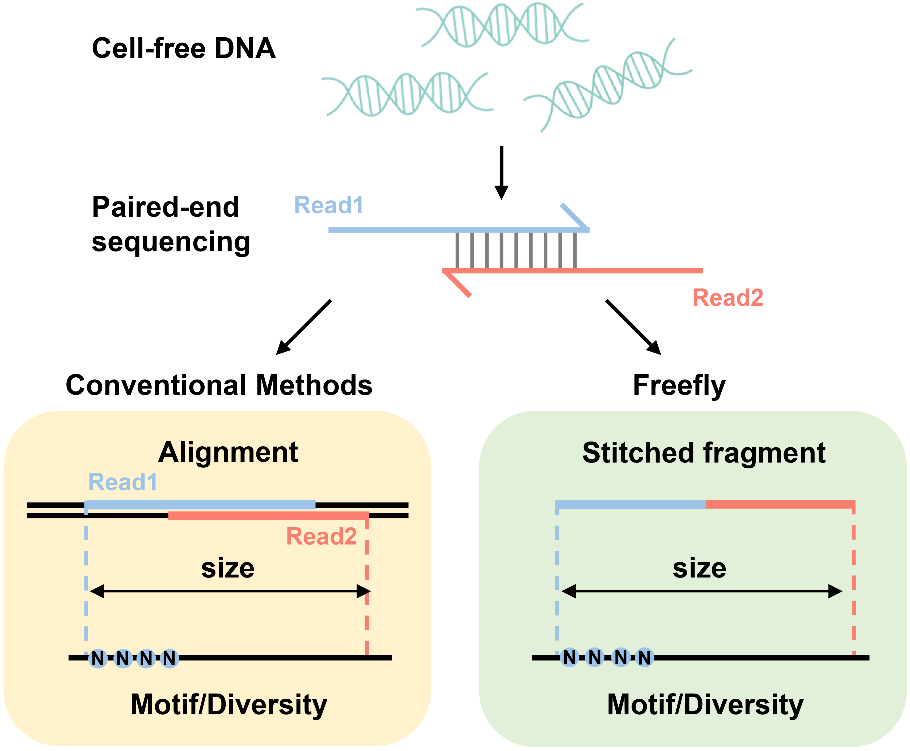
为此,孙坤课题组开发了一款高速无偏cfDNA片段组学分析软件,命名为Freefly。该软件可以在不使用参考基因组的情况下,获得cfDNA片段化组学结果,从而避免了参考基因组带来的偏差,为cfDNA数据分析提供了更好的处理方案,并且在癌症早期诊断等多方面展现了其强大的潜力。
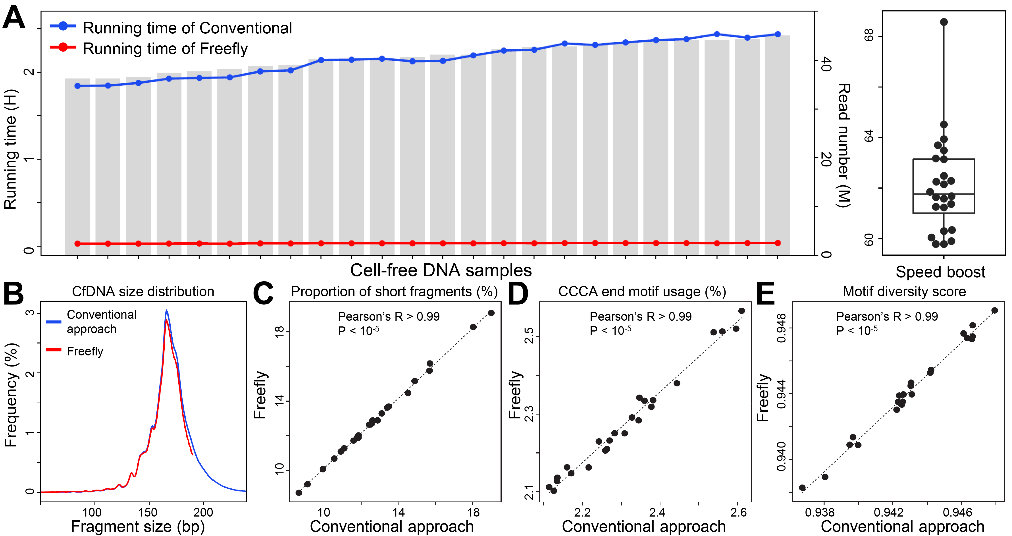
为测试Freefly算法性能表现,该研究使用多组数据来比较传统方法和Freefly性能表现。结果显示,处理50M读数,传统方法获取cfDNA特征值大约需要2小时,而Freefly仅需要2分钟左右即可完成,速度提升了约60倍(如下图所示)。与此同时,Freefly所获得的特征值(Size、CCCA End Motif、以及Motif多样性)与传统方法高度一致,甚至可以提供更好的诊断性能,表明了Freefly在癌症诊断中的价值。
研究支持
本论文所有作者均为深圳湾实验室肿瘤所孙坤课题组成员,其中孙坤博士为本论文的通讯作者,研究助理刘晓懿、联培博士生杨梦琦为本文的共同第一作者。该研究获得国家重点研发计划、广东省基础与应用基础研究基金项目、国家自然科学基金、深圳湾实验室重大项目、深圳湾实验室计算中心平台等支持。
原文链接
https://www.cell.com/cell-reports-methods/fulltext/S2667-2375(24)00149-8
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言
















#cfDNA# #系统偏差#
68